Notiz
Klicken Sie hier , um den vollständigen Beispielcode herunterzuladen
Colormap-Normalisierung #
Objekte, die Colormaps standardmäßig verwenden, ordnen die Farben in der Colormap linear von den Datenwerten vmin bis vmax zu . Zum Beispiel:
wird die Daten in Z linear von -1 bis +1 abbilden, also ergibt Z=0 eine Farbe in der Mitte der Farbtabelle RdBu_r (in diesem Fall weiß).
Matplotlib führt diese Zuordnung in zwei Schritten durch, wobei zuerst eine Normalisierung von den Eingabedaten auf [0, 1] erfolgt und dann auf die Indizes in der Farbtabelle abgebildet wird. Normalisierungen sind im
matplotlib.colors()Modul definierte Klassen. Die standardmäßige lineare Normalisierung ist matplotlib.colors.Normalize().
Künstler, die Daten auf Farbe abbilden, übergeben die Argumente vmin und vmax , um eine matplotlib.colors.Normalize()Instanz zu erstellen, und nennen sie dann:
In [1]: import matplotlib as mpl
In [2]: norm = mpl.colors.Normalize(vmin=-1, vmax=1)
In [3]: norm(0)
Out[3]: 0.5
Es gibt jedoch manchmal Fälle, in denen es nützlich ist, Daten auf nichtlineare Weise Farbkarten zuzuordnen.
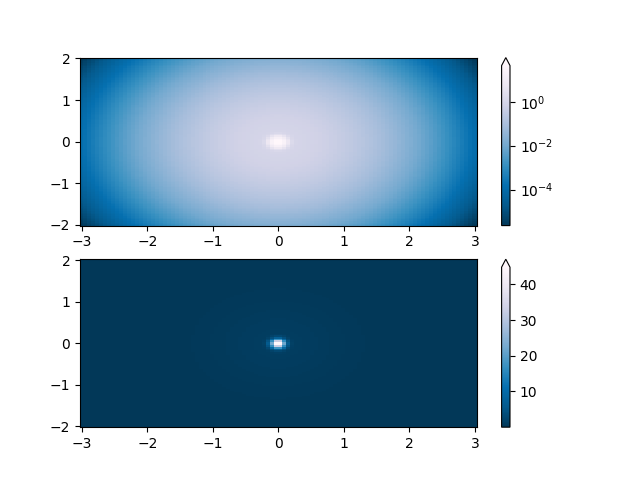
Logarithmisch #
Eine der häufigsten Transformationen besteht darin, Daten durch Logarithmieren (zur Basis 10) darzustellen. Diese Transformation ist nützlich, um Änderungen über unterschiedliche Skalen hinweg anzuzeigen. Mit colors.LogNormnormalisiert die Daten via
\(log_{10}\). Im Beispiel unten gibt es zwei Unebenheiten, eine viel kleiner als die andere. Mit colors.LogNormkönnen die Form und Position jeder Erhebung deutlich gesehen werden:
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.colors as colors
import matplotlib.cbook as cbook
from matplotlib import cm
N = 100
X, Y = np.mgrid[-3:3:complex(0, N), -2:2:complex(0, N)]
# A low hump with a spike coming out of the top right. Needs to have
# z/colour axis on a log scale so we see both hump and spike. linear
# scale only shows the spike.
Z1 = np.exp(-X**2 - Y**2)
Z2 = np.exp(-(X * 10)**2 - (Y * 10)**2)
Z = Z1 + 50 * Z2
fig, ax = plt.subplots(2, 1)
pcm = ax[0].pcolor(X, Y, Z,
norm=colors.LogNorm(vmin=Z.min(), vmax=Z.max()),
cmap='PuBu_r', shading='auto')
fig.colorbar(pcm, ax=ax[0], extend='max')
pcm = ax[1].pcolor(X, Y, Z, cmap='PuBu_r', shading='auto')
fig.colorbar(pcm, ax=ax[1], extend='max')
plt.show()
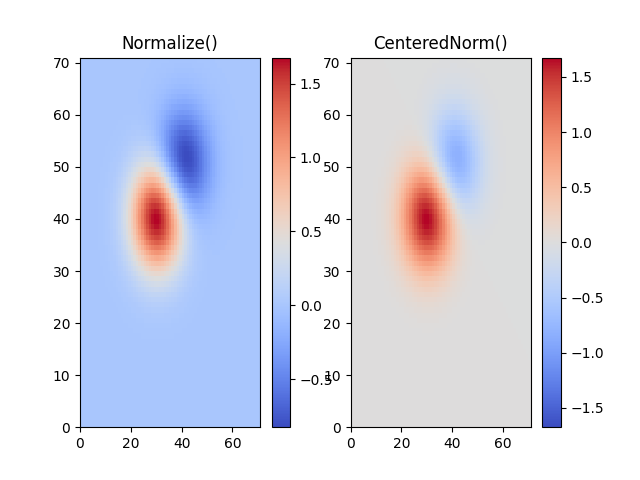
Zentriert #
In vielen Fällen sind Daten symmetrisch um einen Mittelpunkt herum, zum Beispiel positive und negative Anomalien um einen Mittelpunkt 0. In diesem Fall möchten wir, dass der Mittelpunkt auf 0,5 abgebildet wird und der Datenpunkt mit der größten Abweichung vom Mittelpunkt abgebildet wird auf 1,0, wenn sein Wert größer als die Mitte ist, oder auf 0,0 andernfalls. Die Norm colors.CenteredNormerstellt eine solche Abbildung automatisch. Es eignet sich gut für die Kombination mit einer divergenten Farbkarte, die verschiedene Farbkanten verwendet, die sich in der Mitte an einer ungesättigten Farbe treffen.
Wenn das Symmetriezentrum von 0 verschieden ist, kann es mit dem
vcenter- Argument festgelegt werden. Zur logarithmischen Skalierung auf beiden Seiten der Mitte siehe
colors.SymLogNormunten; Um eine andere Zuordnung über und unter der Mitte anzuwenden, verwenden Sie colors.TwoSlopeNormunten.
delta = 0.1
x = np.arange(-3.0, 4.001, delta)
y = np.arange(-4.0, 3.001, delta)
X, Y = np.meshgrid(x, y)
Z1 = np.exp(-X**2 - Y**2)
Z2 = np.exp(-(X - 1)**2 - (Y - 1)**2)
Z = (0.9*Z1 - 0.5*Z2) * 2
# select a divergent colormap
cmap = cm.coolwarm
fig, (ax1, ax2) = plt.subplots(ncols=2)
pc = ax1.pcolormesh(Z, cmap=cmap)
fig.colorbar(pc, ax=ax1)
ax1.set_title('Normalize()')
pc = ax2.pcolormesh(Z, norm=colors.CenteredNorm(), cmap=cmap)
fig.colorbar(pc, ax=ax2)
ax2.set_title('CenteredNorm()')
plt.show()
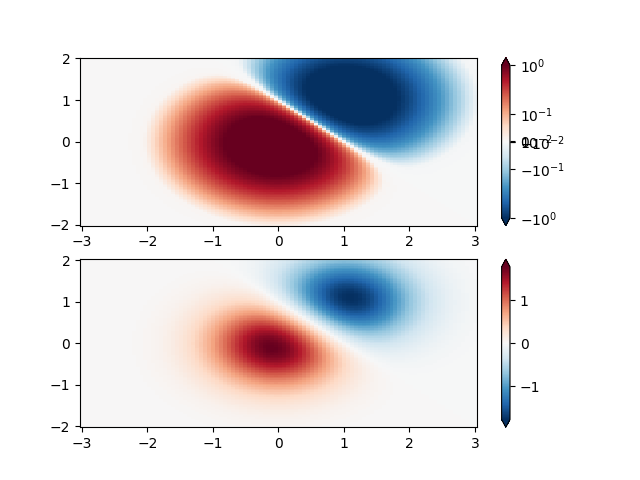
Symmetrisch logarithmisch #
Ebenso kommt es manchmal vor, dass es positive und negative Daten gibt, wir aber trotzdem eine logarithmische Skalierung auf beide anwenden möchten. In diesem Fall werden die negativen Zahlen ebenfalls logarithmisch skaliert und auf kleinere Zahlen abgebildet; B. wenn vmin=-vmax, dann werden die negativen Zahlen von 0 auf 0,5 und die positiven von 0,5 auf 1 abgebildet.
Da der Logarithmus von Werten nahe Null gegen unendlich geht, muss ein kleiner Bereich um Null herum linear abgebildet werden. Mit dem Parameter linthresh kann der Benutzer die Größe dieses Bereichs angeben ( -linthresh , linthresh ). Die Größe dieses Bereichs in der Colormap wird durch linscale festgelegt . Wenn linscale == 1.0 (Standardeinstellung) ist, entspricht der Platz, der für die positive und negative Hälfte des linearen Bereichs verwendet wird, einer Dekade im logarithmischen Bereich.
N = 100
X, Y = np.mgrid[-3:3:complex(0, N), -2:2:complex(0, N)]
Z1 = np.exp(-X**2 - Y**2)
Z2 = np.exp(-(X - 1)**2 - (Y - 1)**2)
Z = (Z1 - Z2) * 2
fig, ax = plt.subplots(2, 1)
pcm = ax[0].pcolormesh(X, Y, Z,
norm=colors.SymLogNorm(linthresh=0.03, linscale=0.03,
vmin=-1.0, vmax=1.0, base=10),
cmap='RdBu_r', shading='auto')
fig.colorbar(pcm, ax=ax[0], extend='both')
pcm = ax[1].pcolormesh(X, Y, Z, cmap='RdBu_r', vmin=-np.max(Z), shading='auto')
fig.colorbar(pcm, ax=ax[1], extend='both')
plt.show()
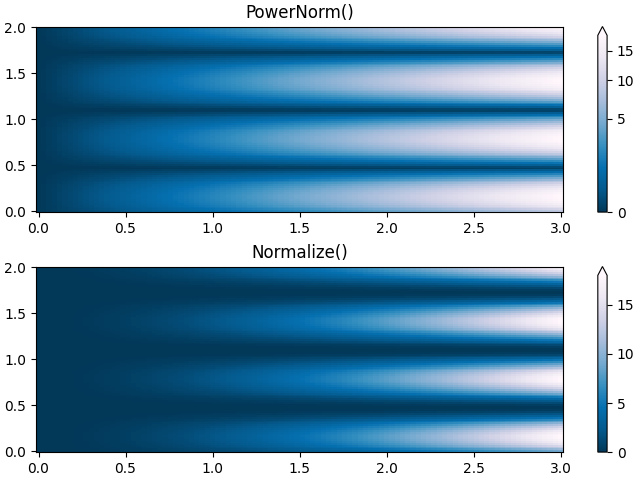
Potenzgesetz #
Manchmal ist es nützlich, die Farben auf eine Potenzgesetzbeziehung neu abzubilden (z\(y=x^{\gamma}\), wo\(\gamma\)ist die Macht). Dazu verwenden wir die colors.PowerNorm. Als Argument wird gamma verwendet ( gamma == 1.0 ergibt nur die standardmäßige lineare Normalisierung):
Notiz
Es sollte wahrscheinlich einen guten Grund geben, die Daten mit dieser Art von Transformation zu plotten. Technische Viewer sind an lineare und logarithmische Achsen und Datentransformationen gewöhnt. Potenzgesetze sind weniger verbreitet, und die Zuschauer sollten ausdrücklich darauf hingewiesen werden, dass sie verwendet wurden.
N = 100
X, Y = np.mgrid[0:3:complex(0, N), 0:2:complex(0, N)]
Z1 = (1 + np.sin(Y * 10.)) * X**2
fig, ax = plt.subplots(2, 1, constrained_layout=True)
pcm = ax[0].pcolormesh(X, Y, Z1, norm=colors.PowerNorm(gamma=0.5),
cmap='PuBu_r', shading='auto')
fig.colorbar(pcm, ax=ax[0], extend='max')
ax[0].set_title('PowerNorm()')
pcm = ax[1].pcolormesh(X, Y, Z1, cmap='PuBu_r', shading='auto')
fig.colorbar(pcm, ax=ax[1], extend='max')
ax[1].set_title('Normalize()')
plt.show()
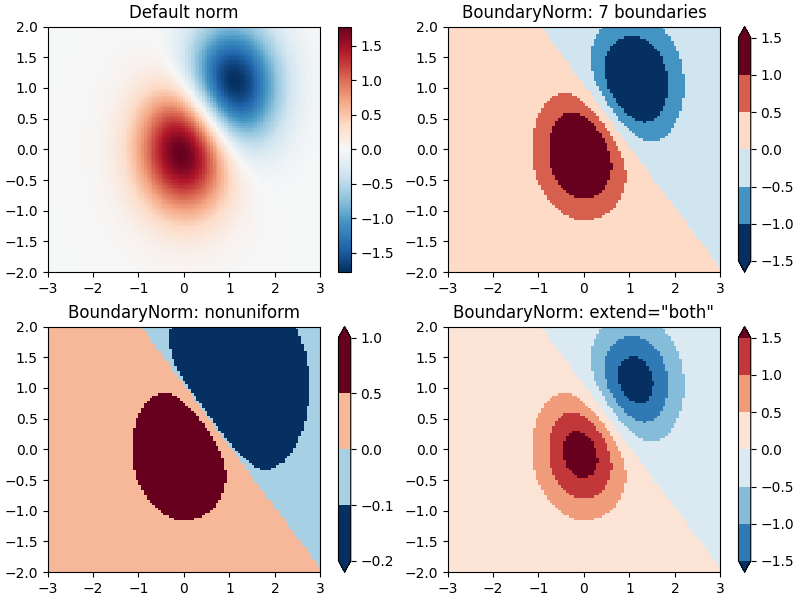
Diskrete Grenzen #
Eine weitere Normalisierung, die mit Matplotlib geliefert wird, ist colors.BoundaryNorm. Zusätzlich zu vmin und vmax nimmt dies als Argumente Grenzen, zwischen denen Daten abgebildet werden sollen. Zwischen diesen "Grenzen" werden dann die Farben linear verteilt. Es kann auch ein Extend - Argument verwenden, um obere und/oder untere Out-of-Bounds-Werte zu dem Bereich hinzuzufügen, über den die Farben verteilt sind. Zum Beispiel:
Hinweis: Im Gegensatz zu den anderen Normen gibt diese Norm Werte von 0 bis ncolors -1 zurück.
N = 100
X, Y = np.meshgrid(np.linspace(-3, 3, N), np.linspace(-2, 2, N))
Z1 = np.exp(-X**2 - Y**2)
Z2 = np.exp(-(X - 1)**2 - (Y - 1)**2)
Z = ((Z1 - Z2) * 2)[:-1, :-1]
fig, ax = plt.subplots(2, 2, figsize=(8, 6), constrained_layout=True)
ax = ax.flatten()
# Default norm:
pcm = ax[0].pcolormesh(X, Y, Z, cmap='RdBu_r')
fig.colorbar(pcm, ax=ax[0], orientation='vertical')
ax[0].set_title('Default norm')
# Even bounds give a contour-like effect:
bounds = np.linspace(-1.5, 1.5, 7)
norm = colors.BoundaryNorm(boundaries=bounds, ncolors=256)
pcm = ax[1].pcolormesh(X, Y, Z, norm=norm, cmap='RdBu_r')
fig.colorbar(pcm, ax=ax[1], extend='both', orientation='vertical')
ax[1].set_title('BoundaryNorm: 7 boundaries')
# Bounds may be unevenly spaced:
bounds = np.array([-0.2, -0.1, 0, 0.5, 1])
norm = colors.BoundaryNorm(boundaries=bounds, ncolors=256)
pcm = ax[2].pcolormesh(X, Y, Z, norm=norm, cmap='RdBu_r')
fig.colorbar(pcm, ax=ax[2], extend='both', orientation='vertical')
ax[2].set_title('BoundaryNorm: nonuniform')
# With out-of-bounds colors:
bounds = np.linspace(-1.5, 1.5, 7)
norm = colors.BoundaryNorm(boundaries=bounds, ncolors=256, extend='both')
pcm = ax[3].pcolormesh(X, Y, Z, norm=norm, cmap='RdBu_r')
# The colorbar inherits the "extend" argument from BoundaryNorm.
fig.colorbar(pcm, ax=ax[3], orientation='vertical')
ax[3].set_title('BoundaryNorm: extend="both"')
plt.show()
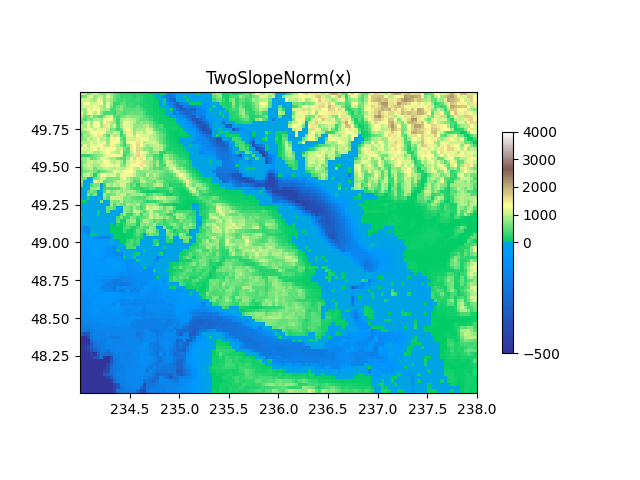
TwoSlopeNorm: Unterschiedliche Zuordnung auf beiden Seiten eines Zentrums #
Manchmal möchten wir auf beiden Seiten eines konzeptuellen Mittelpunkts eine andere Farbtabelle haben, und wir möchten, dass diese beiden Farbtabellen unterschiedliche lineare Skalen haben. Ein Beispiel ist eine topografische Karte, bei der Land und Ozean einen Mittelpunkt bei Null haben, aber Land hat typischerweise einen größeren Höhenbereich als Wasser Tiefenbereich, und sie werden oft durch eine andere Farbkarte dargestellt.
dem = cbook.get_sample_data('topobathy.npz', np_load=True)
topo = dem['topo']
longitude = dem['longitude']
latitude = dem['latitude']
fig, ax = plt.subplots()
# make a colormap that has land and ocean clearly delineated and of the
# same length (256 + 256)
colors_undersea = plt.cm.terrain(np.linspace(0, 0.17, 256))
colors_land = plt.cm.terrain(np.linspace(0.25, 1, 256))
all_colors = np.vstack((colors_undersea, colors_land))
terrain_map = colors.LinearSegmentedColormap.from_list(
'terrain_map', all_colors)
# make the norm: Note the center is offset so that the land has more
# dynamic range:
divnorm = colors.TwoSlopeNorm(vmin=-500., vcenter=0, vmax=4000)
pcm = ax.pcolormesh(longitude, latitude, topo, rasterized=True, norm=divnorm,
cmap=terrain_map, shading='auto')
# Simple geographic plot, set aspect ratio because distance between lines of
# longitude depends on latitude.
ax.set_aspect(1 / np.cos(np.deg2rad(49)))
ax.set_title('TwoSlopeNorm(x)')
cb = fig.colorbar(pcm, shrink=0.6)
cb.set_ticks([-500, 0, 1000, 2000, 3000, 4000])
plt.show()
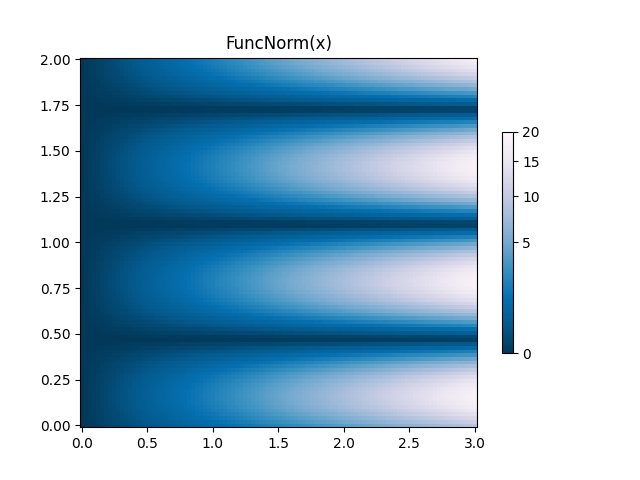
FuncNorm: Normalisierung von Arbiträrfunktionen #
Wenn die oben genannten Normen nicht die gewünschte Normalisierung bieten, können
FuncNormSie mit eigene definieren. Beachten Sie, dass dieses Beispiel dasselbe ist wie PowerNormmit einer Potenz von 0,5:
def _forward(x):
return np.sqrt(x)
def _inverse(x):
return x**2
N = 100
X, Y = np.mgrid[0:3:complex(0, N), 0:2:complex(0, N)]
Z1 = (1 + np.sin(Y * 10.)) * X**2
fig, ax = plt.subplots()
norm = colors.FuncNorm((_forward, _inverse), vmin=0, vmax=20)
pcm = ax.pcolormesh(X, Y, Z1, norm=norm, cmap='PuBu_r', shading='auto')
ax.set_title('FuncNorm(x)')
fig.colorbar(pcm, shrink=0.6)
plt.show()
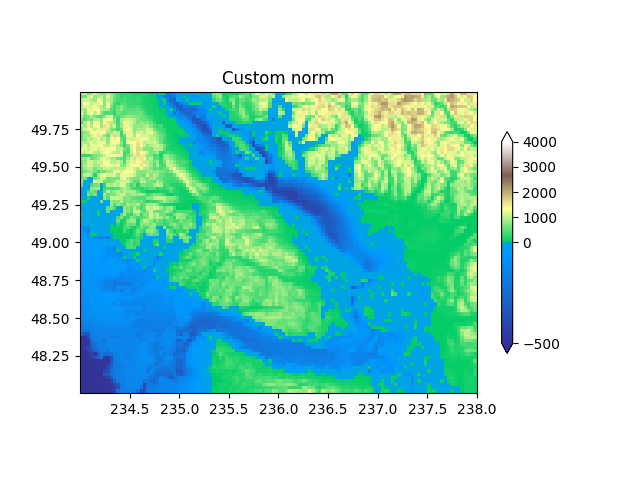
Benutzerdefinierte Normalisierung: Implementieren Sie manuell zwei lineare Bereiche #
Das TwoSlopeNormoben Beschriebene ist ein nützliches Beispiel für die Definition Ihrer eigenen Norm. Hinweis: Damit der Farbbalken funktioniert, müssen Sie eine Umkehrung für Ihre Norm definieren:
class MidpointNormalize(colors.Normalize):
def __init__(self, vmin=None, vmax=None, vcenter=None, clip=False):
self.vcenter = vcenter
super().__init__(vmin, vmax, clip)
def __call__(self, value, clip=None):
# I'm ignoring masked values and all kinds of edge cases to make a
# simple example...
# Note also that we must extrapolate beyond vmin/vmax
x, y = [self.vmin, self.vcenter, self.vmax], [0, 0.5, 1.]
return np.ma.masked_array(np.interp(value, x, y,
left=-np.inf, right=np.inf))
def inverse(self, value):
y, x = [self.vmin, self.vcenter, self.vmax], [0, 0.5, 1]
return np.interp(value, x, y, left=-np.inf, right=np.inf)
fig, ax = plt.subplots()
midnorm = MidpointNormalize(vmin=-500., vcenter=0, vmax=4000)
pcm = ax.pcolormesh(longitude, latitude, topo, rasterized=True, norm=midnorm,
cmap=terrain_map, shading='auto')
ax.set_aspect(1 / np.cos(np.deg2rad(49)))
ax.set_title('Custom norm')
cb = fig.colorbar(pcm, shrink=0.6, extend='both')
cb.set_ticks([-500, 0, 1000, 2000, 3000, 4000])
plt.show()
Gesamtlaufzeit des Skripts: ( 0 Minuten 5.849 Sekunden)